One Tip Per Day: Learning notes for Unix, Perl, R, HTML, Javascript, Google API and mostly Bioinformatics……【阅读全文】
Tag Archives: R
[荐]第二代测序技术数据分析教程与视频
在线教程(Fall 2012 – Analyzing High Throughput Sequencing Data):http://www.personal.psu.edu/iua1/pages/fall-2012-bmmb-597D.html
在线视频(新一代测序技术数据分析[英语]):http://www.youku.com/playlist_show/id_17418799_ascending_1_mode_pic_page_1.html……【阅读全文】
[转载]关于统计-生物学家需要知道的五件事
全文提纲
- Non parametric statistics.
- R (or I guess S).
- The problem of multiple testing, and how to handle it, either with the Expected value, or FDR, and the backstop of many of piece of bioinformatics – large scale permutation.
- The relationship between Pvalue, Effect size, and Sample size.
- Linear models and PCA.
……【阅读全文】
人类基因组中的基因密度、特征长度和SNP密度
一、源起
本周,在为本科生设计生物信息学实验的时候,突发奇想,想要利用Galaxy工具计算一下人类基因组的常识性信息,如:每条染色体上的基因密度,外显子、内含子等特征(feature)的平均长度,SNP在UTR、编码区、内含子等不同特征区域的密度。
二、工具
系统平台:Linux(Ubuntu 12.04,AMD64)。
UCSC Table:下载基因组数据至本地。
Galaxy:用于在线处理基因组数据。
BEDTools(v2.16.2):用于本地处理基因组数据
R(v2.15.1):绘制图表。
其他:Vim(v7.3.429)。
三、数据库
human genome:hg19
dbSNP:135
四、结果
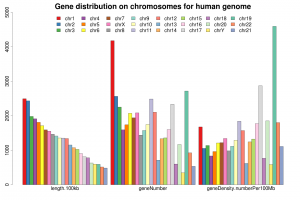
1.基因在每条染色体上的数目与密度。
- 数据表格
| chromosome | length.bp | length.100kb | geneNumber | geneDensity.numberPer100Mb |
|---|---|---|---|---|
| chr1 | 249250621 | 2492.50621 | 4177 | 1675.82330718 |
| chr2 | 243199373 | 2431.99373 | 2563 | 1053.86784858 |
| chr3 | 198022430 | 1980.2243 | 2251 | 1136.73991376 |
| chr4 | 191154276 | 1911.54276 | 1592 | 832.835149343 |
| chr5 | 180915260 | 1809.1526 | 1739 | 961.223503203 |
| chr6 | 171115067 | 1711.15067 | 2071 | 1210.29669468 |
| chr7 | 159138663 | 1591.38663 | 1940 | 1219.06264853 |
| chrX | 155270560 | 1552.7056 | 2083 | 1341.5292635 |
| chr8 | 146364022 | 1463.64022 | 1437 | 981.79865541 |
| chr9 | 141213431 | 1412.13431 | 1573 | 1113.91670669 |
| chr10 | 135534747 | 1355.34747 | 1749 | 1290.4439922 |
| chr11 | 135006516 | 1350.06516 | 2485 | 1840.65189861 |
| chr12 | 133851895 | 1338.51895 | 2102 | 1570.39241021 |
| chr13 | 115169878 | 1151.69878 | 711 | 617.348921738 |
| chr14 | 107349540 | 1073.4954 | 1334 | 1242.66950748 |
| chr15 | 102531392 | 1025.31392 | 1357 | 1323.49710028 |
| chr16 | 90354753 | 903.54753 | 1600 | 1770.79782399 |
| chr17 | 81195210 | 811.9521 | 2331 | 2870.85900757 |
| chr18 | 78077248 | 780.77248 | 599 | 767.188925511 |
| chr20 | 63025520 | 630.2552 | 1167 | 1851.63089491 |
| chrY | 59373566 | 593.73566 | 347 | 584.435167664 |
| chr19 | 59128983 | 591.28983 | 2716 | 4593.34807095 |
| chr22 | 51304566 | 513.04566 | 924 | 1801.00929028 |
| chr21 | 48129895 | 481.29895 | 534 | 1109.49753786 |
- 条形图展示
Continue reading
关于EMBOSS和Galaxy的生物信息学实验课程设计
本人设计的针对本科生的生物信息学实验课程设计,包括涉及EMBOSS和Galaxy的三个题目:
- 基于 EMBOSS 平台对 *** 基因进行序列分析
- 基于 Galaxy 平台分析 *** 物种基因在基因组中的分布
- 基于 Galaxy 平台分析 *** 物种 SNP 在不同特征区域中的分布
对于每一个题目,我都记录了详细的操作步骤,对于想学习EMBOSS和Galaxy(主要是后者)的朋友可能会有所帮助。
Continue reading
[荐]Short Course 2010 Documentation
在R中绘制韦恩图的几种方法
-
使用limma包
1 2 3 4 5 6 7 8 9 10 11 12 13 14 | #安装limma包 #source("http://www.bioconductor.org/biocLite.R") #biocLite("limma") #载入limma包 library(limma) hsb2 《- read.table("http://www.ats.ucla.edu/stat/R/notes/hsb2.csv", sep=',', header=T) attach(hsb2) hw 《- (write《=60) hm 《- (math 《=60) hr 《- (read 《=60) c3 《- cbind(hw, hm, hr) a 《- vennCounts(c3) vennDiagram(a, include = "both", names = c("High Writing", "High Math", "High Reading"), cex = 1, counts.col = "red") |
-
使用gplots包
1 2 3 4 5 6 7 8 9 10 11 12 13 | #安装gplots包 #install.packages("gplots") #载入gplots包 library(gplots) oneName 《- function() paste(sample(LETTERS,5,replace=TRUE),collapse="") geneNames 《- replicate(1000, oneName()) GroupA 《- sample(geneNames, 400, replace=FALSE) GroupB 《- sample(geneNames, 750, replace=FALSE) GroupC 《- sample(geneNames, 250, replace=FALSE) GroupD 《- sample(geneNames, 300, replace=FALSE) input 《-list(GroupA,GroupB,GroupC,GroupD) venn(input) |
[续]生成特定碱基比例的随机序列
前文《生成特定碱基比例的随机序列》介绍了如何用Perl来生成特定碱基比例的随机序列。……【阅读全文】
情人节特献之“我心永恒”
-
极坐标中的心形[R]
1 2 3 4 | library(plotrix) t《-seq(-pi,pi,0.1) r《-1-cos(t/2) radial.plot(r,t,rp.type="p",lwd=3,line.col=2) |
-
2D的中国心[R]
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 | #heart n=30000 p=sort(runif(n,min=0,max=pi)) p=sin(p)+rnorm(n) plot(sort(rnorm(n)),-p,col="red",pch="·",axes = F, ylab="",xlab=paste("By 微微 ",Sys.Date()),main="Happy Birthday") #stars p《-c() q《-c() rr《-c(0.68,rep(0.32,4)) #locator()定的下面坐标,精确但不准确。 m《-c(-1.2511927,-0.7707085,-0.5538095,-0.6129638,-0.8293381) n《-c(-0.079779159,0.42312375,0.05632304,-0.45719796,-0.8440031) for (k in c(1:5)) { r=rr[k] for (i in 1:5) { p[2*i-1]=sin(pi*18/90)/sin(pi*54/90)*r*sin(pi*(i*72+36)/90) q[2*i-1]=sin(pi*18/90)/sin(pi*54/90)*r*cos(pi*(i*72+36)/90) p[2*i]=sin(pi*18/90)/sin(pi*54/90)*0.38*r*sin(pi*(i*72+18)/90) q[2*i]=sin(pi*18/90)/sin(pi*54/90)*0.38*r*cos(pi*(i*72+18)/90) } x=c(p[1],p[2],p[5],p[6],p[9],p[10],p[3],p[4],p[7],p[8],p[1]) y=c(q[1],q[2],q[5],q[6],q[9],q[10],q[3],q[4],q[7],q[8],q[1]) x=x+m[k] y=y+n[k] polygon(x, y, col="yellow",border = "yellow") } |
-
3D的中国心[R]
预览:

代码(下载):mychina
-
浪漫的表白
在R中同时生成多种格式的图片
0700c16fc091a7c9b7cb70fd740d764c005……【阅读全文】